摘要:Seahorse实时细胞能量代谢分析系统的检测数据受到孔间细胞数目、细胞活性和细胞在每个孔中均匀程度的影响,需要进行归一化处理得到有效数据后再进行分析。Seahorse细胞能量代谢分析数据归一化有以下几种常用方法:基于总蛋白定量或总DNA定量的间接评估法,以及基于细胞计数的评估法。基于成像的细胞计数方法是指利用显微成像系统对已经进行过细胞能量代谢测试过的特殊细胞板孔中细胞进行成像,然后使用合适参数对图像进行细胞数目的定量分析。相对于总蛋白定量和总DNA定量的间接评估法,活细胞显微成像计数法操作简单快速,更直接更容易实现自动化;而且在检测细胞数目的同时可区分活/死细胞,可消除实验过程中因药物处理浓度或时间不同导致的孔间细胞活性状态差异而引入的代谢数据差异;同时对细胞种植的均匀程度都也有一定的直观参考价值。本文将介绍如何利用成像仪器对Seahorse XFe24 实验板内的各孔细胞进行准确计数。
关键词: 显微成像, 细胞计数, 归一化, 细胞能量代谢 (Seahorse XFe24)
材料与试剂
- Seahorse XFe24 FluxPak实验板 (Agilent, Seahorse, catalog number: 102342-100)
- Hoechst33342 (ThermoFisher, Invitrogen, catalog number: H3570)
- Propidium Iodide (PI) (ThermoFisher, Invitrogen, catalog number: P3566)
- 75%酒精
仪器设备
- 水平板式离心机 (Eppendorf, model: Centrifuge 5910 R)
- 恒温培养箱 (无二氧化碳)
- 2.5 µl、1000 µl移液枪
- 细胞成像多功能检测仪 (PerkinElmer公司, model: EnSight 以及 BioTek公司, model: Cytation 5)
实验步骤
一、细胞染色 (Hoechst 33342 染色和PI染色)
- 从细胞能量代谢分析仪中取出检测板,弃去上层,保留下层细胞板和板盖 (图1)。

图1. 从细胞能量代谢分析系统中取出的检测板 A. 上下两层,弃去上层;B. 下层细胞板正面,红色叉处为空白孔,固定不铺设细胞。
- 计算细胞板内已有的检测培养基的体积,按照1:1000加入Hoechst 33342及PI核染色试剂。一般情况细胞板里含有750 µl检测培养基,分别加入Hoechst 33342及PI各0.75 µl,盖上细胞板板盖,手动轻摇板子混匀。(Anal Chem)
- 200 × g 室温下离心 2 min。
- 放入培养箱37 °C孵育 15 min。
注:检查培养箱中的水盘,为有水状态,保持培养箱中恒温恒湿。
二、图像采集
本文将分别阐述使用两台细胞成像多功能检测仪器 (Perkin Elmer公司的Ensight及BioTek公司的Cytation 5) 进行图像采集及定量分析的流程。Seahorse XFe24实验板的尺寸参数见图2,对该板成像前需先在成像仪器中设置好板型及相应参数。其中“Bottom Elevation” 可根据实际拍照的清晰层面来确定。
注:成像前,用75%酒精喷湿无尘擦拭纸,擦拭细胞板底部的外表面,去除细胞板板盖。 图2. Seahorse XFe24实验板参数设置 (界面来自BioTek公司的Cytation 5)
图2. Seahorse XFe24实验板参数设置 (界面来自BioTek公司的Cytation 5) - Ensight多功能成像酶标仪成像方法
通道选择及成像参数 (见图3):Hoechst 33342染料检测用385 nm-extended/413-498 nm (Blue);PI染料检测用525 nm/555-604 nm (Orange)。
注:也可同时增加一个明场检测通道作为辅助参考。

图3. Ensight成像参数设置
- Cytation 5多功能成像酶标仪图像收集方法
通道选择如下:Hoechst 33342染料检测用 365 nm LED Ex377/50 Em447/60;PI染料检测用 523 nm LED Ex531/40 Em647/57 (图4)。使用Cytation 5 可通过每孔拍摄4个视野捕获整孔信息 (图4)。聚焦方式可根据硬件配置情况,优先选择"Laser autofocus" (图4B)。

图4. Cytation 5成像参数设置
结果与分析
一、成像结果及图像分析方法设定
图像定量分析思路如下:我们首先根据Hoechst 33342染色圈选细胞核位置,由于Hoechst 33342可同时进入活、死细胞,因此被圈选的“object”同时包括了活细胞及死细胞,由此得到细胞总数;然后分析各圈选“object”区域PI通道的荧光强度;通过设置PI通道荧光强度的阈值,分选出死细胞亚群 (PI+ cells),软件会自动对该亚群进行计数;Hoechst 33342阳性的细胞数减去PI染色阳性的细胞数,即为孔内实际存活细胞的数目。
注:有部分PI染色阳性的细胞,其Hoechst 33342通道的信号非常弱,在object圈选过程中没有被选中,不进入整个分析过程,由于这类细胞属于死细胞,因此对结果的分析准确性无影响。
以下将分别介绍Ensight及Cytation两台仪器进行图像分析的参数设置方法:
- Ensight仪器软件参数的设置方法:
- Cytation图像分析参数的设置方法
为更好地对图像进行定量分析,Cytation 5可对拍摄的图片进行去除背景、视野拼接等预处理。我们首先对图片进行“image preprocessing”提高图像信噪比之后,对一个孔拍摄的4个视野进行拼接 (image stitching), 得到整孔拼接图片 (图7),之后进入后续定量分析步骤 (图8)。
注:图片拼接有个参数非常重要,应该无损失拼接,不要选择默认值。
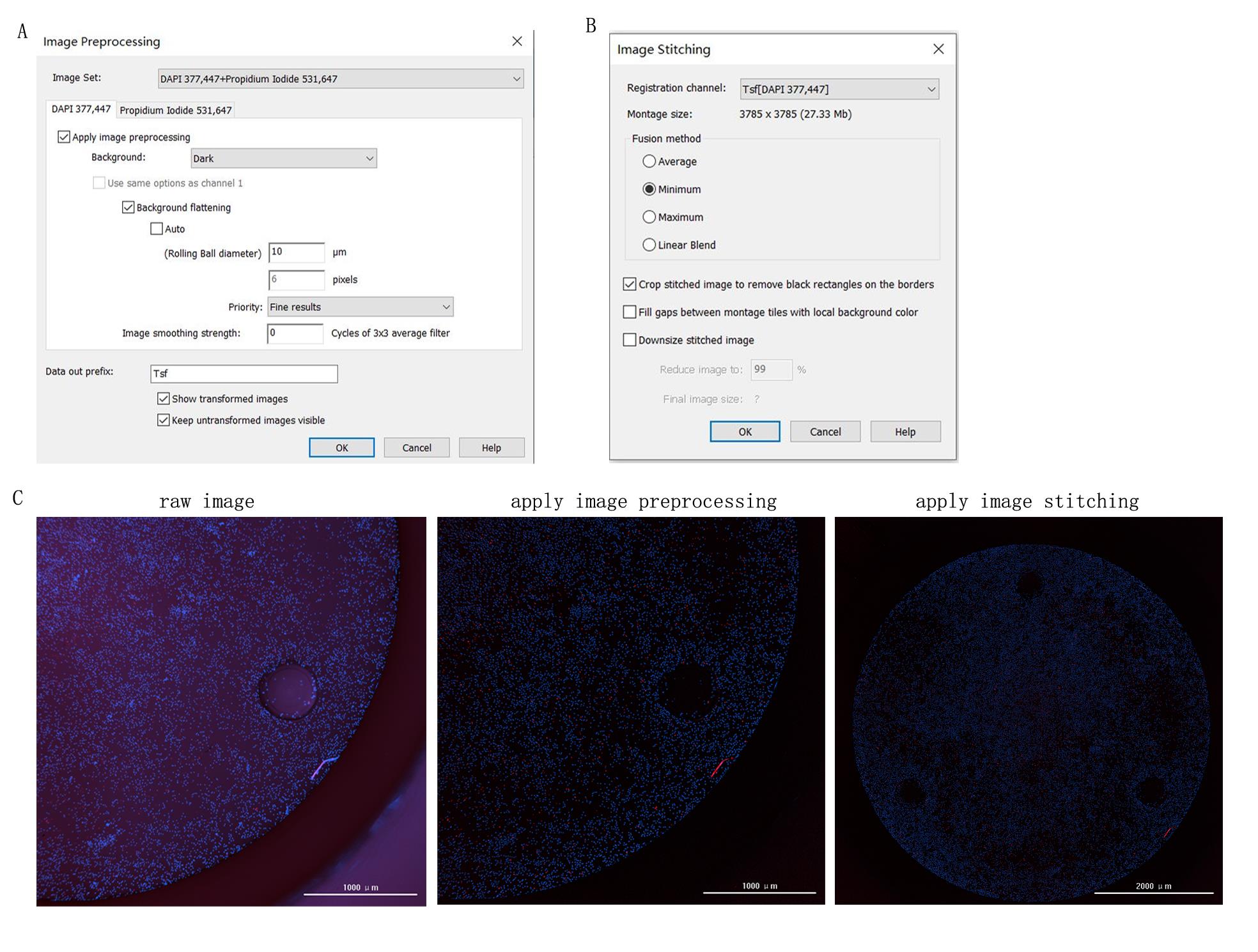
图7. Cytation原始图片及分析前图像预处理
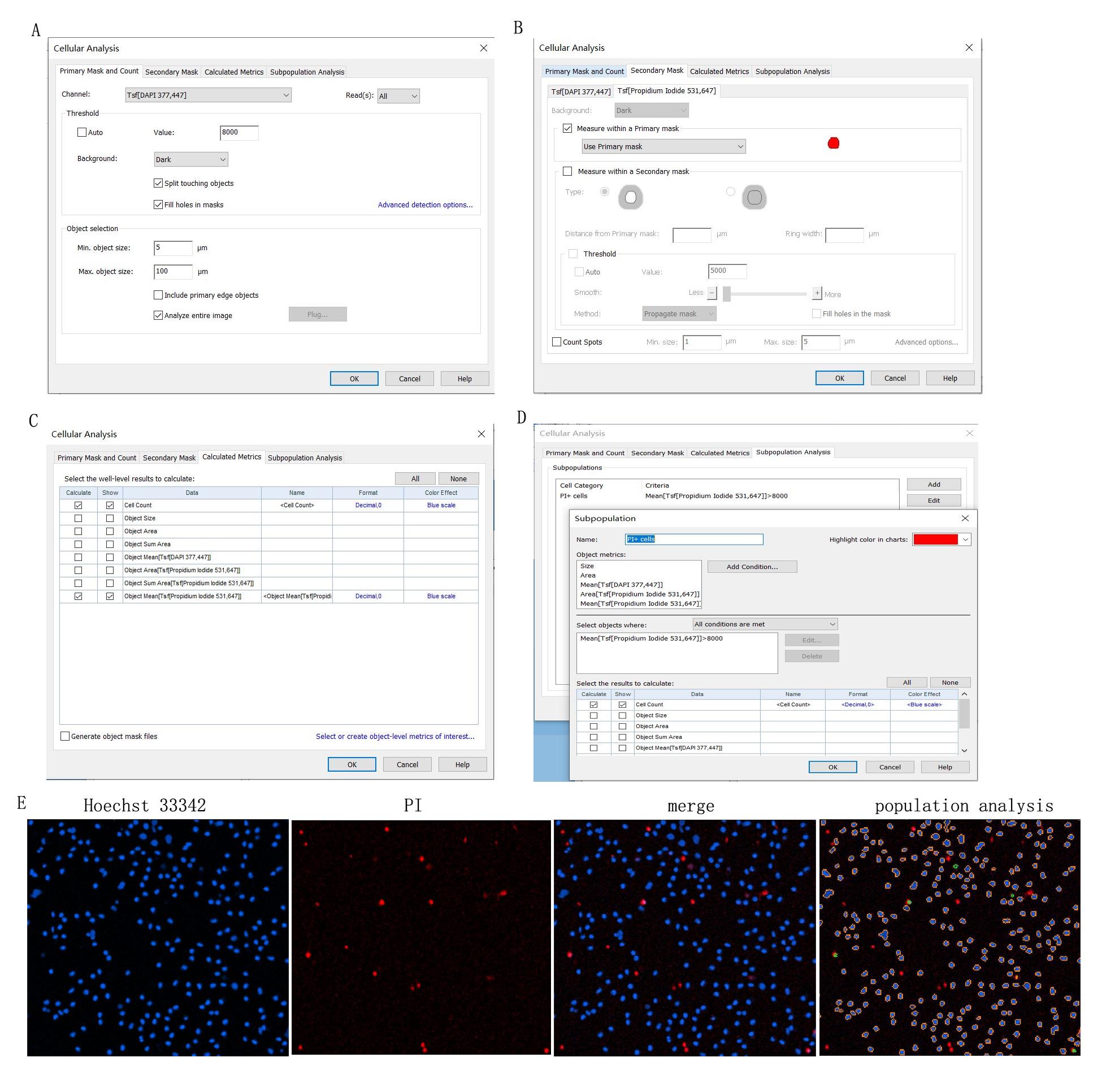
图8. Cytation 5多功能成像酶标仪分析方法及分析效果图 A. 设定Hoechst 33342的阈值进行总细胞数圈选;B. 在Hoechst33342圈选的区域计算每个细胞核PI通道的荧光强度;C. 选定分析需要定量的参数;D. 设置PI荧光强度的阈值作为确定PI+细胞的判断标准;E. 各通道图片及分析过程细胞核圈选情况,橙色为所有Hoechst 33342阳性的细胞 (用来计算总细胞数),绿色圈选为Hoechst33342及PI双阳性的细胞群体 (用来计算死细胞数)。
二、Seahorse实验结果归一化
为了验证上述所设定的图像定量分析方法是否准确合理,我们将HeCaT野生型细胞株(B细胞) 及某基因敲除的细胞株 (A细胞) 分别以不同密度接种至板内,进行Seahorse实验后,染核,利用上述方法进行成像并进行细胞数定量分析。
利用如下公式对各孔数据进行细胞数归一化处理,如图9输入活细胞数目:Normalized OCR = OCR / (Hoechst 33342阳性细胞数 – PI 阳性细胞数)* 比例因子。Seahorse仪器得到的原始数据值如图10,OCR值受细胞种类及细胞接种数影响显著。从图11可以看出,利用此方法进行数据归一化处理后(J Cell Mol Med),成功消除了孔间细胞数不同导致的数据差异,可反应细胞能量代谢的实际差异。

图9. 输入活细胞数到Seahorse软件中相应的位置,可以自动处理
注:比例因子Factor通常设置为细胞平均数或取整,并在Unit处修改对应数Cells,其只影响Y轴数值的倍数。

图10. A类细胞和B类细胞分别用两种浓度种植在Seahorse24孔细胞板内所呈现的细胞能量代谢检测结果

图11. A类细胞和B类细胞经过细胞计数归一化处理后所呈现的细胞能量代谢检测结果
失败经验
- 细胞孔内不要引入小颗粒,由于是基于细胞成像的图片质量分析细胞计数的,小颗粒或不溶物可能会被染料附着识别计入。操作过程中培养基是现配现用的,并且是在开放式空间操作检测的,非常容易长细菌,不易放置过长时间。另外染色液如需稀释,需新鲜配置,或使用前离心取上清,移液管等都用干净无杂质的。
- 控制合适的细胞密度。细胞密度过高,贴在板底的细胞不是单层,造成细胞核重叠,无法准确分辨细胞个数,分析出的细胞数将会不准确。
- 由于Seahorse实验要求单层细胞贴壁状态,核染料浓度按照说明书使用即可,但不排除个别种类细胞对染料浓度有特殊需要,可适当增加染色时间或染色剂量'。
- Seahorse实验结束后,细胞状态会受到影响,导致细胞贴壁不牢。细胞染色过程中,为避免细胞从板底脱落,操作要格外小心轻柔,或者预先用多聚-L-赖氨酸 (常用)、Cell-Tak细胞组织粘合剂、Matrilgel基质胶、Collagen鼠尾I型胶原蛋白(干细胞常用)等试剂辅助包被细胞板板底。
- 经过辅助贴壁处理的悬浮细胞也可用这种方法进行归一化处理,但悬浮细胞如果粘连,将无法准确分辨细胞个数,分析出的细胞数将会不准确,要求均匀独立铺在细胞板底。另外悬浮细胞一般个体小于贴壁细胞,如果4倍镜细胞成像分辨率不够,分析出的细胞数将会不准确,可以考虑用更高倍数成像。
- 个别种类细胞经过Seahorse实验的三至四种抑制剂处理后,细胞活性会降低,细胞成像计数实验要快速完成,不然死细胞的计数会比真实情况偏多。
致谢
感谢中国科学院分子细胞科学卓越创新中心分子生物学技术平台和化学生物学技术平台全体成员的建议和帮助。
参考文献
- Silva, L. P., Lorenzi, L. P., Purwaha, P., Yong, V., Hawke, D. H. and Weinstein, J. N. (2013). Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Anal Chem 85(20): 9536-9542.
- Quent, V. M. C., Loessner, D., Friis, T., Reichert, J. C. and Hutmacher, D. W. (2010). Discrepancies between metabolic activity and DNA content as tool to assess cell proliferation in cancer research. J Cell Mol Med 14(4): 1003-1013.
Please login or register for free to view full text
View full text
Download PDF
Q&A
Copyright: © 2021 The Authors; exclusive licensee Bio-protocol LLC.
引用格式:赵曚, 田维新, 韩帅, 郭蕴郦, 陈铭. (2021). 基于成像的细胞计数方法---Seahorse细胞能量代谢分析 系统数据归一化方法. // 高内涵成像及分析实验手册.
Bio-101: e1010856. DOI:
10.21769/BioProtoc.1010856.
 How to cite:
How to cite: Zhao, M., Tian, W. X., Han, S., Guo, Y. L. and Chen, M. (2021). Image-based Cell Number Analysis---Data Normalization Method in Seahorse Cell Metabolic Analysis. // High-Content Imaging and Analysis Protocol eBook.
Bio-101: e1010856. DOI:
10.21769/BioProtoc.1010856.
